近日,上海科技大学免疫化学研究所白芳课题组在国际学术期刊Journal of Cheminformatics上发表题为“DeepSA: A Deep-learning Driven Predictor of Compound Synthesis Accessibility ”的研究论文,该工作设计了一个名为DeepSA的化学语言模型,用于化合物合成可及性预测。
随着ChatGPT和LLM的蓬勃发展,以分子生成模型为代表的一系列AIGC算法不断涌现,为生物医药等各个领域注入了新活力。分子生成模型是一种新颖的从头药物设计方法,不仅可以帮助药物化学家加快对于化学空间的搜索效率,也能够降低药物发现的投入消耗。近年来,基于深度学习的分子生成模型不断出现,然而,这些模型所生成分子的结构有时过于复杂,导致合成难度过大,即分子合成可及性低,从而阻碍了药物的开发进程。因此,在药物发现早期对于分子可进行准确评估,可减少分子合成过程中投入,加速药物研发。本研究基于化学语言模型,发展了高效、高准确性的化合物合成可及性评估方法DeepSA(如图1)。
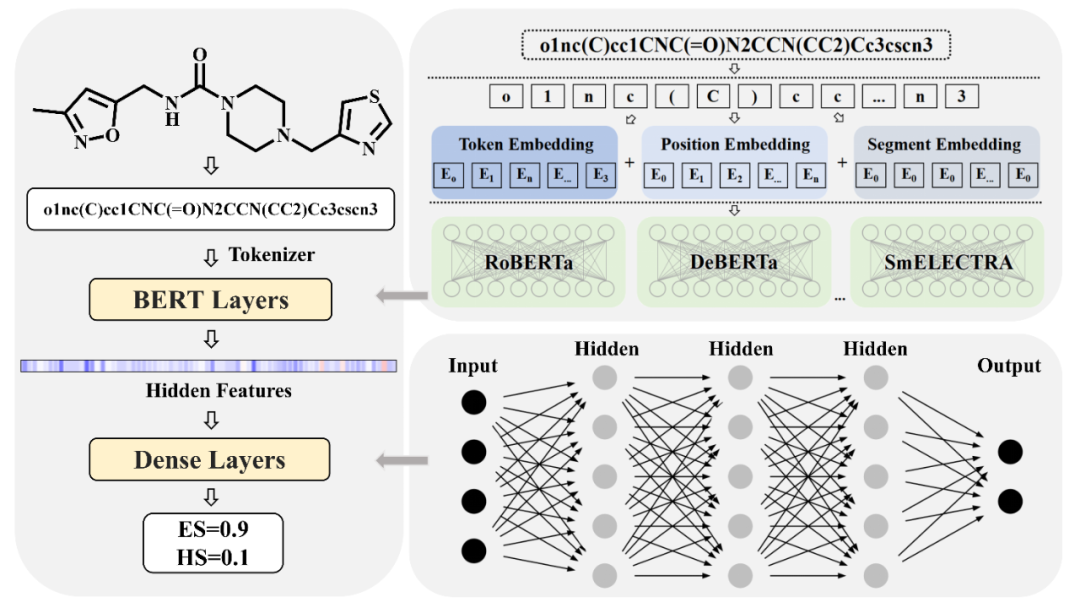
图1. DeepSA的模型框架
用于训练DeepSA模型的数据来源于ZINC,ChEMBL,GDB等公共数据库中的80万个分子,通过逆合成分析算法得到了每个分子的合成难度标签,合成步数大于十的分子被视为难合成分子,反之则为易合成。分子通过SMILES编码输入模型之中,借鉴自然语言处理领域的经验,利用基于Transformer的多种预训练语言模型(RoBERTa,DeBERTa, ELECTRA等)对分子在token和position层面进行embedding处理,提取分子特征,之后利用多层感知机进行特征解码,输出该分子的合成可及性得分。DeepSA在较大规模的具有实际合成路线的分子中进行了检验,其性能优于其他现有合成可及性预测算法,并具有优良的鲁棒性。同时,DeepSA在识别难以合成的分子方面具有很高的早期富集率,可以帮助用户准确选择分子进行合成。
作者提供了该算法的在线预测服务,部署在课题组网站中(https://bailab.siais.shanghaitech.edu.cn/deepsa),方便用户使用,模型的训练和推理源代码也可在Github中进行获取(https://github.com/Shihang-Wang-58/DeepSA),方便相关研究人员使用或更改模型,以满足个性需求。作者认为,DeepSA可以作为计算化学领域中分子生成模型的优秀过滤器,提升分子生成的性能。
免化所白芳课题组2022级硕士研究生王世航和2021级博士研究生王林为论文共同第一作者,白芳研究员为论文的通讯作者。上海科技大学为第一完成单位。该项工作得到了科技部重点研发计划、临港实验室、国家自然科学基金委、上海市科委和上海市教委项目(上海市生物大分子与精准医药前沿科学研究基地等)的支持。该项工作也得到iHuman研究所程建军研究员和上科大高性能计算平台的大力支持。
论文链接:https://doi.org/10.1186/s13321-023-00771-3





